1. 発表者:

高尾 大輔(東京大学大学院医学系研究科 助教)
長尾 幸子(東京大学薬学部 学部生:研究当時)
坂本 美佳(情報・システム研究機構国立遺伝学研究所 特任研究員)
知念 拓実(東京大学大学院薬学系研究科 助教)
岡田 康志(東京大学国際高等研究所ニューロインテリジェンス国際研究機構(WPI-IRCN)主任研究者/東京大学大学院医学系研究科・大学院理学系研究科 教授/理化学研究所生命機能科学研究センター(BDR)チームリーダー)
2.発表のポイント:
◆ DNA染色やオルガネラの染色画像だけを使ってディープラーニングにより細胞周期を判定するAIツールの開発に成功した。
◆ 細胞周期を判定する過程でAIが発見した情報を使って研究者が解析することで、細胞周期によって変動する特徴量を画像から抽出するアプローチを確立した。
◆ 仮説や思い込みに左右されずに画像から未知の情報を発掘することが可能になり、顕微鏡を使った細胞・発生生物学研究から画像診断などの医療応用に至るまで、幅広い分野の技術革新への貢献が期待できる。
3.発表概要:
東京大学大学院医学系研究科の高尾大輔 助教と東京大学薬学部の長尾幸子 学部生(研究当時)の研究チームは、国立遺伝学研究所の坂本美佳 特任研究員、東京大学大学院薬学系研究科の知念拓実 助教、東京大学国際高等研究所ニューロインテリジェンス国際研究機構(WPI-IRCN)の岡田康志 主任研究者(東京大学大学院医学系研究科・大学院理学系研究科 教授、理化学研究所生命機能科学研究センター(BDR)チームリーダー)らと共同で、ディープラーニングと定量解析により細胞画像から未知の情報を抽出する技術の開発に成功しました。大量の細胞画像から一つ一つの細胞を自動的に切り出し、ディープラーニングにより細胞周期などを判定するAIツールと、その情報をさらに解析することで、細胞周期によって変動する核の形状やゴルジ体の配置パターンなどを抽出する技術を確立しました。これは、人間の目ではとらえにくいわずかな細胞内の変動をAIが発見し、研究者に教えてくれる技術です。
細胞周期によって細胞内の組成や構造は大きく変わることが予想されますが、実際に何がどのように変動するのかを顕微鏡画像から網羅的かつ定量的に解析するためのアプローチは限られていました。具体的に細胞内の何に着目すればいいのかを知るために、今回、共同研究チームはディープラーニングを使って、細胞周期によって変動するパラメータを画像の中から見つける技術を開発しました。これまで研究者が「なんとなく」「経験的に」とらえていた現象や、そもそも見過ごされていた情報を、AIの手助けにより発掘・定量化しようという試みです。その結果、本研究で開発したAIは、DNAやゴルジ体の染色画像の中から、細胞周期によってわずかに変動する特徴を発見しました。この情報を使って共同研究チームが画像を詳しく解析したところ、核やゴルジ体の面積などの特定のパラメータを測定することで画像から細胞周期を分類できることが分かりました。すなわち「画像のどこに着目すれば目的の情報が得られるのかAIが教えてくれる」という研究サポートツールの開発に成功しました。
本研究で開発したツールは汎用性が高く、他の多くの研究への応用を想定しています。細胞周期以外の判定はもちろん、細胞画像以外の画像データへの応用も可能なため、細胞・発生生物学から医療画像の解析など、基礎から応用まで幅広い分野の研究をサポートする強力なツールとなることが期待されます。
本研究の成果は、2020年4月22日に米国細胞生物学会誌Molecular Biology of the Cellに掲載されます。
4.発表内容:
(1)研究の背景と経緯
細胞内の物質の組成や構造は、細胞周期を通じてダイナミックに変動します(図1)。最も顕著な変動は、分裂期における紡錘体の形成や細胞分裂です。一方、間期においてもDNA の複製やオルガネラの増加など、分裂期に向けて細胞内ではダイナミックな変化が起こっています。しかし、これらは、顕微鏡観察で捉えるにはあまりにも小さな変動です。このようなごくわずかな変動を顕微鏡画像の中から検出することは可能でしょうか。近年注目を浴びるディープラーニングを応用することで、この課題に挑みました。
(2)研究の内容
本研究では、固定後に抗体等を用いて染色したヒトおよびマウスの培養細胞の蛍光顕微鏡画像を用いて、細胞周期などを判定しました。特に本研究で中心となるのは、DNAやゴルジ体をそれぞれ単独あるいは組み合わせて染色し、間期の細胞を画像からG1/S期とG2期に分類する課題です。そのために、画像分類に多用される畳み込みニューラルネットワーク(注1)を使ったディープラーニングにより、細胞画像を細胞周期によって分類するAIツールを開発しました(図2)。多くの細胞を含む大きな画像から個々の細胞を認識して自動的に切り出し、細胞周期を判定することができます。ここで使われている独自のPython(注2)コードは、論文公開に合わせてGitHubで公開されています。
上記AIツールによる画像分類を検証した結果、DNAの染色画像単独、あるいはゴルジ体または微小管プラス端の染色画像との組み合わせにより、90%以上の精度で正しくG1/S期とG2期の細胞を分類することに成功しました(図3A, B)。さらにこのとき、AIは画像の中のどこに注目して細胞周期を判定しているのか、Grad-CAM解析(注3)により可視化しました。その結果、例えばDNAとゴルジ体の組み合わせ画像を用いて学習させたモデルでは、G1/S期だと判定したときにはゴルジ体の輪郭を、G2期だと判定したときは加えて核全体をAIは注目していることが分かりました(図3C)。このような結果を基に、例えばDNAの染色の強さ(明るさ)とゴルジ体の面積を同時に2次元にプロットしてみると、G1/S期とG2期の細胞をうまく分離できることが分かりました(図3D)。さらに興味深いことに、AIの判定の自信の強さともいえるSoftmax(注4)の値は、両グループの境界部分で小さくなり、すなわちこの2次元プロット上で判別が難しい領域はAIも判定結果にあまり自信がないというきれいな結果となりました。
これらの解析での重要な結論は、次の2点です。1)細胞周期の分類など、潜在的に画像に含まれる情報を使って画像を分類したいとき、画像のどこに着目すればいいのかを教えてくれるAIツールを開発しました。2)細胞周期にともなうわずかな細胞内の構造の変動など、画像の中に潜在的に含まれていながらも人の目では気づきにくい情報を、AIのサポートにより発掘するアプローチを確立しました。これは、仮説や先入観にとらわれにくい、データ駆動型のアプローチです。細胞画像の中から特定の構造やパターンを選び出したりノイズを除去して画像を復元するためにAIを活用する研究は多くありますが、本研究ではそこからさらに踏み込んで、細胞画像の中に隠された生物学的に重要な情報を取り出すことに成功しました。
(3)今後の展開
本研究で開発したAIツールの利点の一つは、その汎用性にあります。他のオルガネラや細胞内構造、細胞周期以外の対象へのアプローチの拡充はもちろん、使用するデータは画像データであれば顕微鏡画像である必要性もありません。よって、細胞・発生生物学分野の多くの研究や診断ツールなど医療分野への応用など、幅広い分野の発展への貢献が期待できます。また、今回AIが発見した現象は、これまでの知見によって簡単に説明できる、研究者にも納得しやすいものでした。今後は研究者が想像すらしていないような現象の発見につながる研究発展が期待されます。
本研究は、科学技術振興機構(JST)戦略的創造研究推進事業 チーム型研究(CREST)「統合1細胞解析のための革新的技術基盤」(研究総括:菅野 純夫 東京医科歯科大学 非常勤講師)における「超解像3次元ライブイメージングによるゲノムDNAの構造、エピゲノム状態、転写因子動態の経時的計測と操作」(研究代表者:岡田 康志)および文部科学省世界トップレベル研究拠点プログラム 東京大学国際高等研究所ニューロインテリジェンス国際研究機構(WPI-IRCN)の支援のもとで行われたものです。
また、本研究の一部は以下の事業による支援を受けて行われました。
● 文部科学省 科学研究費 新学術領域研究「学術研究支援基盤形成」先端バイオイメージング支援プラットフォーム(ABiS)
● 文部科学省 科学研究費 新学術領域研究「情報物理学でひもとく生命の秩序と設計原理」
● 日本学術振興会科学研究費基盤研究(C)「分子ダイナミクスの定量・操作と数理モデリングによる中心体複製メカニズムの理解」
● 武田科学振興財団
● 島津科学振興財団
● コニカミノルタ科学技術振興財団
5.発表雑誌:
雑誌名:Molecular Biology of the Cell(オンライン版:4月22日)
論文タイトル:Robust Classification of Cell Cycle Phase and Biological Feature Extraction by Image-Based Deep Learning
著者:Yukiko Nagao, Mika Sakamoto, Takumi Chinen, Yasushi Okada, Daisuke Takao
DOI番号:https://doi.org/10.1091/mbc.e20-03-0187
6.問い合わせ先:
【研究に関する問い合わせ先】
東京大学大学院医学系研究科
助教 高尾 大輔(たかお だいすけ)
東京大学国際高等研究所ニューロインテリジェンス国際研究機構(WPI-IRCN)主任研究者/
東京大学大学院医学系研究科・大学院理学系研究科 教授/
理化学研究所生命機能科学研究センター(BDR) チームリーダー
岡田 康志(おかだ やすし)
【広報担当者問い合わせ先】
東京大学国際高等研究所ニューロインテリジェンス国際研究機構(WPI-IRCN)広報担当
佐竹 真由紀(さたけ まゆき)
E-mail:pr@ircn.jp
理化学研究所 広報室 報道担当
TEL:048-467-9272
E-mail:ex-press@riken.jp
7.用語解説:
(注1)畳み込みニューラルネットワーク(CNN):ディープラーニングアルゴリズムの一つ。画像を扱うのが得意なため、画像の分類に多用される。多数のフィルターを使って畳み込み(convolution)と呼ばれる演算で画像を処理する畳み込み層を多層に重ねたモデル構造を持つ。
(注2)Python:プログラミング言語の一種。ディープラーニングを含む機械学習アルゴリズム実装のための公開パッケージ(ライブラリー)が豊富に提供され、関連分野ではスタンダードとして使われている。
(注3)Grad-CAM解析:学習済みの畳み込みニューラルネットワークにおいて、入力画像のどの部分が判定結果に大きな影響を与えているかを可視化するための解析。結果をヒートマップで出力できるため、AIモデルの構造を視覚的に解釈することができる。
(注4)Softmax:ニューラルネットワークの出力層に多用される活性化関数の一つ。0と1の間の値を取る滑らかな曲線を描く関数で、出力層にある複数の項目のSoftmax値を足し合わせると1になる。例えば出力層のSoftmax値が(G1/S, G2) = (0.8, 0.2)であれば判定結果は”G1/S”となり、(G1/S, G2) = (0.1, 0.9)であれば判定結果は”G2”となる。(G1/S, G2) = (0.4, 0.6)であれば判定結果は”G2”であるが、ほぼ半々に近い微妙な結果とも言える。そのためここではSoftmaxの値をAIモデルの自信の強さと表現している。
8.添付資料:
図1 細胞周期と細胞内の変化
大きく細胞内構造が変化するM期(分裂期)に比べ間期(G1~G2期)の細胞内の変動は一見すると明瞭ではない。しかし、実際には多くのダイナミックな変動が起こっているはずである。

図2 AIモデルの模式図
畳み込みニューラルネットワークを使ったディープラーニングモデルを開発した。
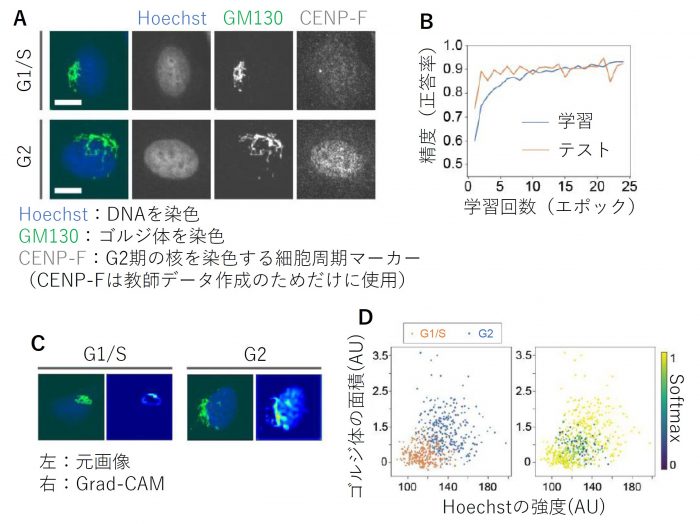
図3 細胞周期の分類と特徴量抽出
(A) DNAとゴルジ体を染色した画像の例。CENP-Fの画像はAIモデルには見せない。スケールバーは10μm。
(B) 学習の例。このモデルでは精度91%を達成。
(C) Grad-CAM解析によってAIモデルが画像のどこに着目したかを可視化。
(D) 元画像から2つのパラメータを選んで定量した後、2次元にプロットした例。細胞周期で色分けしたプロット(左)とSoftmaxの値で色分けしたプロット(右)を表示している。


